Разработана обновленная классификация CRISPR-Cas, учитывающая открытия последних пяти лет. Она включает 2 класса, 7 типов и 46 подтипов, тогда как в предыдущем исследовании 2020 года было выделено 6 типов и 33 подтипа. Ранее неописанный тип VII встречается в основном у архей, относится к классу 1 и содержит новую нуклеазу Cas14. Некоторые новые варианты типа V, имеющие Cas12 с инактивированным нуклеазным доменом, вероятно, способны ингибировать репликацию мишени без разрезания.
Credit:
123rf.com
Даже для большинства биологов CRISPR-Cas — это прежде всего CRISPR-Cas9, система, широко применяемая в экспериментах, а теперь и в терапевтическом редактировании. Однако это всего лишь один из вариантов, существующих в природе, который был обнаружен у Streptococcus pyogenes и принадлежит к типу II класса 2. Известное нам разнообразие систем CRISPR-Cas велико, и оно продолжает расширяться. С учетом новых открытий разработана обновленная эволюционная классификация CRISPR-Cas. Среди соавторов статьи, опубликованной в Nature Microbiology, — Евгений Кунин с коллегами, Дженнифер Дудна, Эммануэль Шарпантье, Фэн Чжан, Филипп Хорват, Виргиниюс Шикшнис и другие известные исследователи CRISPR.
Системы CRISPR-Cas отличаются количеством белковых элементов, вовлеченных в разные этапы «адаптивного иммунного ответа» бактерии на бактериофаг или мобильный элемент: интеграцию нового спейсера в CRISPR, экспрессию элементов, необходимых для уничтожения фага при повторном контакте с ним, собственно интерференцию (взаимодействие с мишенью и ее уничтожение). Так, если системы класса 1 включают несколько белков Cas, необходимых для процессинга pre-crРНК, связывания с crРНК и мишенью и ее последующего расщепления, то в системах класса 2 различные функции можно объединять один Cas-белок. (Одна из причин, по которым практические применения нашли прежде всего системы, включающие Cas9, а также Cas12 и Cas13: использовать в качестве инструмента один белок проще, чем несколько.)
Классификация систем CRISPR-Cas, основанная, насколько это возможно, на эволюционных связях, имеет важное значение для поиска и характеристики CRISPR-Cas-локусов в новых геномах бактерий и архей. Однако системы CRISPR–Cas не имеют универсальных маркеров, подходящих для филогенетического анализа, а организация локусов CRISPR-Cas и сами белки Cas быстро эволюционируют, что осложняет построение классификации.
Для создания предыдущих версий классификации (последняя из них была опубликована в 2020 году) использовали как сравнение архитектуры и состава генов локусов CRISPR-Сas, так и кластеризацию по сходству последовательностей и филогенетический анализ консервативных белков Cas, например, интегразы Cas1, играющая ключевую роль во встраивании спейсеров.
В исследовании 2020 года было выделено 6 типов и 33 подтипа CRISPR-Сas. С тех пор был открыт седьмой тип и 13 ранее неизвестных подтипов, таким образом, обновленная классификация включает 2 класса, 7 типов и 46 подтипов.
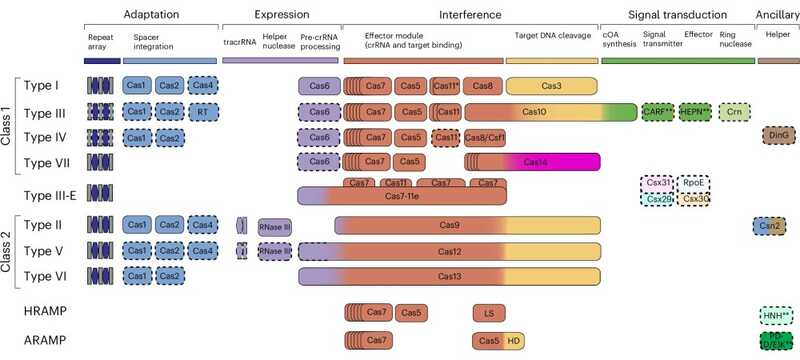
Существующие системы CRISPR-Cas: обобщенная схема. Credit: Nat Microbiol (2025). DOI: 10.1038/s41564-025-02180-8 | CC BY 4.0
Анализ распространенности вариантов CRISPR-Cas в геномах и метагеномах показывает, что выявленные в последние пять лет варианты достаточно редки по сравнению с известными ранее системами: новые типы и подтипы составляют всего 0,3% репертуара CRISPR-Cas в полностью секвенированных прокариотических геномах. Эти малораспространенные варианты составляют «длинный хвост» распределения CRISPR-Cas у прокариот и еще не охарактеризованы экспериментально.
К ранее неописанному типу VII принадлежат системы CRISPR-Cas, обнаруженные в основном в нескольких геномах таксономически разнообразных архей. Они отнесены к классу 1 и включают нуклеазу Cas14, которая состоит из металло-β-лактамазы (β-CASP) и домена, гомологичного C-концу Cas10, что говорит об эволюционной связи между типами III и VII.
Два новых подтипа, принадлежащих к типу III, — III-G и III-H — потеряли способность синтезировать циклические олигоаденилаты (cOA). Таким образом, они утратили зависимый от cOA сигнальный путь, который индуцирует коллатеральную РНКазную активность (способность расщеплять другие молекулы, кроме мишени) в большинстве систем типа III. Есть признаки, указывающие на то, что системы этих подтипов могут расщеплять ДНК-мишени. Еще один новый подтип этого типа, III-I, скорее всего, расщепляет РНК.
Системы CRISPR-Cas типа V (нуклеазы Cas12, используемые в том числе для создания тест-систем) замечательны широким разнообразием действия. Они способны распознавать и расщеплять одноцепочечные РНК и ДНК (Cas12g), или же таргетирование РНК может сопровождаться побочным расщеплением одноцепочечной РНК, одноцепочечной и двухцепочечной ДНК (Cas12a2). Но кроме того, существуют независимо возникшие в ходе эволюции варианты Cas12 с инактивированным нуклеазным доменом. По крайней мере некоторые из них могут подавлять репликацию мишени, не разрезая ее — например, связываясь с мишенью под управлением crRNA и блокируя ее транскрипцию.
Авторы также обсуждают другие новые необычные функции CRISPR-Cas, в частности, варианты типа IV, расщепляющие целевую ДНК (ранее для типа IV было показано только ингибирование транскрипции мобильных элементов).
Кроме того, открытие ранее неизвестных систем CRISPR-Cas углубляет понимание их эволюции. Авторы перечисляют следующие ее тенденции: взаимодействие между структурным и функциональным усложнением и редукционистской, упорядочивающей эволюцией; «запутанность» эволюции CRISPR-Cas и различных типов мобильных элементов, в соответствии с концепцией guns for hire, когда, например, нуклеазы мобильных элементов становятся частью защитной системы; использование систем CRISPR-Cas для других функций, не связанных с защитой, таких как индукция клеточной смерти; перетасовка эффекторных модулей (ответственных за таргетирование мишени) и приобретение вспомогательных генов. При этом эволюционные траектории классов 1 и 2, по-видимому, существенно различаются, равно как и траектории эффекторных и адаптационных (ответственных за включение интеграцию новых спейсеров) модулей систем CRISPR-Cas.
Источник
Makarova, K.S., et al. An updated evolutionary classification of CRISPR–Cas systems including rare variants // Nature Microbiology (2025). DOI: 10.1038/s41564-025-02180-8